Newsletter – 20 de agosto 2024
Trabalho em pauta: Exploring SoySNP50K and USDA Germplasm Collection Data to Find New QTLs Associated with Protein and Oil Content in Brazilian Genotypes
Autores: Jessica Nayara Basílio Silva, Rafael Delmond Bueno, Teresinha de Jesus Feitosa de Sousa, Yan Pablo Moreira Xavier, Luiz Claudio Costa Silva, Newton Deniz Piovesan, Cleberson Ribeiro, Maximiller Dal-Bianco.
A domesticação da soja (Glycine max) resultou na perda de diversidade genética, com 50% da diversidade genética e 81% dos alelos raros perdidos. Para criação de novas cultivares são utilizados pequenos conjuntos de linhagens com intuito de gerar maior rendimento, em contrapartida, ocasionou a redução do pool genético disponível. Muitos programas de melhoramento têm como objetivo identificar regiões genômicas relacionadas ao teor de óleo e proteína nas sementes de soja, sendo já relatado na literatura, efeitos significativos associados em duas regiões nos cromossomos 15 e 20. Apesar de serem encontradas grande quantidade de dados sobre marcadores associados a essas características, informações acerca do teor de proteína encontrado nas cultivares utilizadas em países produtores do grão são escassas. Estudos relacionados a QTLs associados aos teores de óleo e proteína presentes nas cultivares elite, podem acelerar significativamente o desenvolvimento de novas cultivares com estas características. Com isso, o objetivo deste trabalho foi identificar SNPs responsivas à acumulação de proteína e óleo em sementes de soja, para servirem como potenciais alvos para uso em programas de melhoramento do Brasil. Como objetivo também visa compreender a influência dos ancestrais Roanoke, Lee e Bragg nas variedades brasileiras.
Foi conduzido um estudo de associação genômica ampla (GWAS) utilizando dados públicos de fenotipagem e genotipagem de Bandillo et al. (2015). Além dos dados deste estudo, foram adicionados 34 genótipos obtidos do banco de germoplasma do Programa de Melhoramento da Qualidade da Soja (PMQS/UFV). Os dados dos genótipos foram recuperados utilizando https://www.soybase.org/snps/download.php , exceto os 34 genótipos citados.
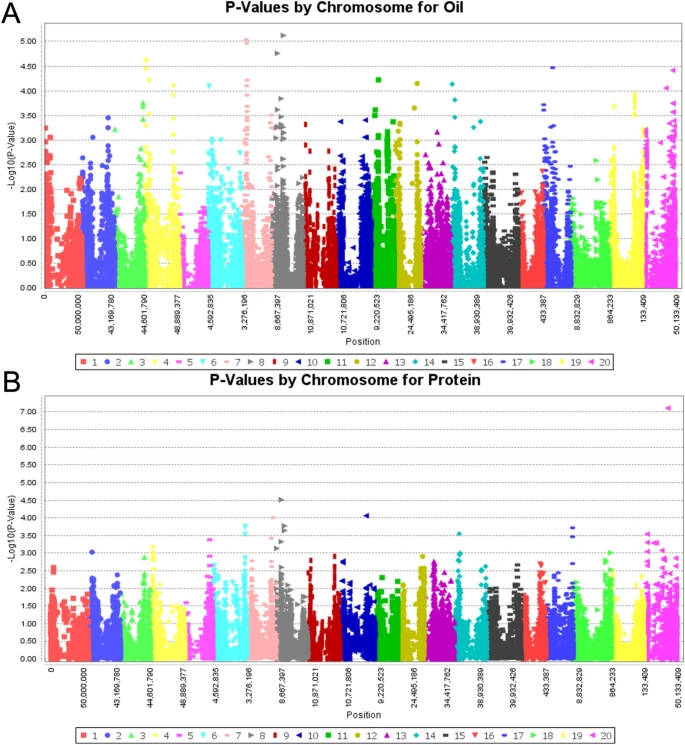
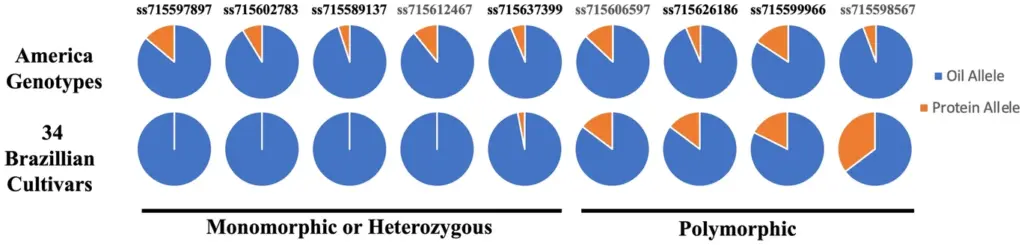
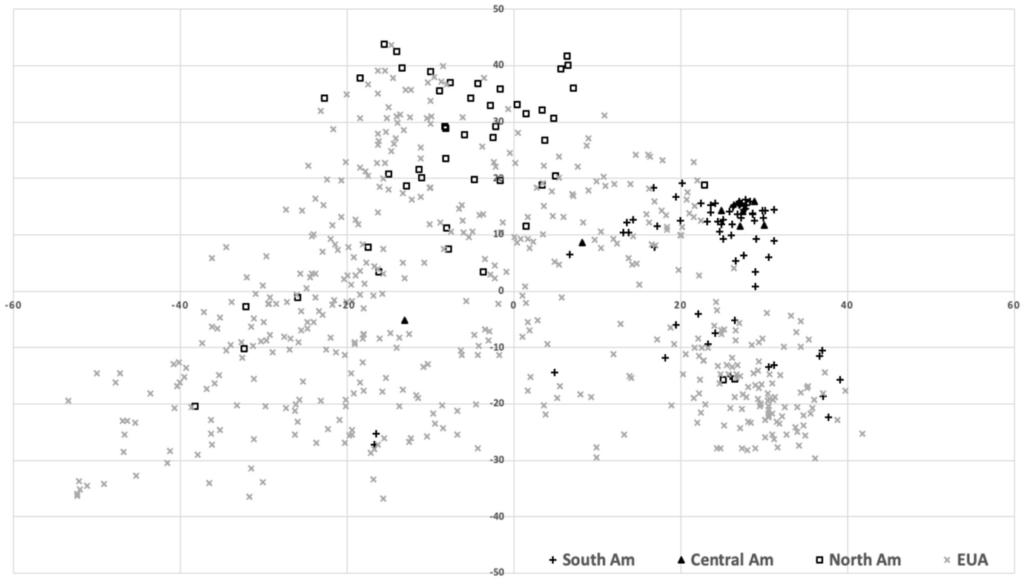
Para a genotipagem, utilizou-se a metodologia KASP, e a fenotipagem foi feita com espectrometria de infravermelho próximo (NIR). Os dados foram analisados com o software TASSEL para PCA, agrupamento filogenético e GWAS. A avaliação dos efeitos dos SNPs no teor de proteína e óleo incluiu comparações estatísticas usando teste t e determinação do efeito aditivo com os programas GENES, GQMOL e TASSEL 5.0. A análise de 604 genótipos mostrou uma sub-representação de genótipos brasileiros em estudos de GWAS, com apenas 15 genótipos brasileiros incluídos. Uma árvore filogenética revelou quatro clusters distintos de genótipos brasileiros e ancestrais dos EUA. Certos QTLs importantes para proteína e óleo são raros em genótipos brasileiros. Foram identificados 22 novos SNPs associados ao teor de proteína e óleo, com R^2 variando de 2,428% a 4,859%. Dentre estes marcadores alguns exibiram efeitos aditivos consideráveis em proteínas (ss715597897, ss715606597 e ss715637399) e óleo (ss715589137, ss715598567, ss715602783, ss715599966, ss715612467, 617832 e ss715626186). Das 22 QTLs identificadas foram selecionadas 9 SNPs para testar a hipotese de que sua presença pode ser valiosa em programas de melhoramento. Sendo assim, foram genotipadas as 34 cultivares do germoplasma do programa, verificando que dois eram monomórficos e quatro eram polimórficos, sendo que as frequências alélicas destes últimos são mantidas ou crescentes. Estes marcadores podem ser facilmente utilizados em programas de melhoramento que visam aumentar o teor de proteína ou óleo, sendo os dois mais promissores ss715606597 e ss715598567.
Confira o trabalho completo em: 10.1007/s10528-024-10698-5
Resumo por: Brizza Fernandes dos Santos Vargas
Deixe um comentário
Você precisa fazer o login para publicar um comentário.